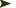Accueil
/ Dossiers / Autres dossiers / Génétique (Produire plus, produire mieux 4/6) : prouesses et promesses / Gagner du temps avec la sélection assistée par marqueurs
Accueil
/ Dossiers / Autres dossiers / Génétique (Produire plus, produire mieux 4/6) : prouesses et promesses / Gagner du temps avec la sélection assistée par marqueurs
Génétique (Produire plus, produire mieux 4/6)
Prouesses et promesses

La pression sociétale contraint les sélectionneurs dans les domaines végétal et animal à accélérer l’intégration de nouveaux critères dans leurs programmes. La génomique s’annonce comme l’outil incontournable pour répondre à cette demande et améliorer l’efficacité de la sélection.
Article 3 :
Gagner du temps avec la sélection assistée par marqueurs
Mise au point dans les années 1980, la sélection assistée par marqueur (Sam) est aujourd’hui utilisée en routine chez les principaux semenciers et les trois principales races laitières élevées en France (prim’holstein, montbéliarde et normande). Elle améliore l’efficacité de la sélection.
Des «bornes» dans le génome
Du côté végétal, il fallait, par exemple, au moins sept rétrocroisements (croisement d’un individu avec l’un de ses parents) pour développer une variété avec la sélection classique, qui est basée sur l’étude des phénotypes (aspect des individus). Il n’en faut désormais plus que trois ou quatre. La technique consiste à mettre en évidence et suivre la transmission, de génération en génération, de régions chromosomiques, les QTL. Ces dernières sont associées à des caractères quantitatifs que l’on cherche à sélectionner: la précocité, l’efficience de la nutrition azotée, la résistance à une maladie ou au stress hydrique chez les plantes, la production laitière, les taux butyreux et protéique, la fertilité, les cellules, le développement de la mamelle chez les vaches…
Pour détecter et localiser ces QTL, le sélectionneur utilise des marqueurs moléculaires équivalents à des «bornes» placées le long des chromosomes, à proximité du QTL. Le choix des meilleurs individus est donc plus précis. Cela facilite aussi la sélection de variétés cumulant des critères agronomiques complexes.
 La Sam est actuellement en phase d’accélération avec le développement de marqueurs de «nouvelle génération», les SNP (A l'heure de la génomique
).
La Sam est actuellement en phase d’accélération avec le développement de marqueurs de «nouvelle génération», les SNP (A l'heure de la génomique
).
Grâce à eux, à la fin de 2008, prim’holsteins, normandes et montbéliardes entreront dans une nouvelle ère: celle de la Sam 2. Ces trois races travailleront sur quinze caractères à l’aide de 1.152 marqueurs au lieu des huit caractères de la Sam 1, avec ses 45 marqueurs. «Avec la Sam 1, nous expliquions entre 30 et 50% de la variabilité génétique, détaille Alain Malafosse, ancien directeur adjoint de l’Union des coopératives d’élevage et d’insémination animale (Unceia). Avec la Sam 2, nous arriverons entre 60 et 70%.»
Chez les végétaux, «on peut aussi expliquer entre 50 et 70% de la variabilité génétique variétale avec les QTL identifiés par les marqueurs moléculaires, explique André Gallais d’AgroParisTech. Le reste est du ressort de la sélection phénotypique.»
dossier réalisé par Isabelle Escoffier et Eric Roussel (extrait de La France agricole n° 3232, du 25 avril 2008)
météo agricole
Vendredi 18 décembreLes températures maximales :

cours & marchés
LES PLUS LUS